Phylogenetic tree
Who are the ancestors of the dinosaurs?
Science 1994 Nov 18;266(5188):1229-1232
Compare a dinosaur's sequence of mitochondrial cytochrome b with other 12 species, Human, Dog, Rabbit, Rhinoceros, Dugong, Mouse, Whale, Bovine, Sicklebill, Chicken, Magpie, and Frog. (ClustalX)
* Create a phylogenetic tree to analyze their relationship. (Phylip3.5c)A sequence of dinosaur's mitochondrial cytochrome b:มี
cccttctattattcattctcattctattcgttattcttgtactccacacatccaaacaacaaagcataatattccacccattgagtccattccta
tcctgattcttagtccccgaaccttttacactcacatg
มีขั้นตอนการทำ ดังนี้
1. หาลำดับเบส(sequence)ที่เป็น mitochondrial cytochrome b ของสิ่งมีชีวิตทั้ง 12 ชนิด จาก NCBI และบันทึกไว้เป็น FASTA format ไว้ใน txt file

2.ทำ Sequence alignment ด้วยโปรแกรม ClustalX จะได้ output file ที่ชื่อ dnd file และ phylip file

3. เปลี่ยนชื่อ phy file ให้เป็น infile เพื่อใช้ในโปรแกรม phylip 3.5c
4. เปิดโปรแกรมที่ชื่อ seqboot.exe เพื่อหาไฟล์ที่ชื่อ infile จากนั้นโปรแกรม seqboot จะทำหน้าที่สร้างชุดซ้ำของข้อมูลหรือ bootstrapping เพื่อนำไปใช้ในการสร้าง phylogenetic tree

5.หลังจากที่โปรแกรม seqboot ทำการสร้างชุดซ้ำของข้อมูลจะได้ output เป็น outfile
6.ภายหลังจากที่ได้ outfile ให้ทำการเปลี่ยนชื่อ infile ที่เรานำเข้าตอนแรกเพื่อที่จะได้เปลี่ยน outfile เป็น infile ใหม่ (เพราะ outfile ที่เปลี่ยนเป็น infile ใหม่นี้จะมีขนาดเพิ่มขึ้นจากขบวนการ bootstrapping )

7.เลือก squboot แล้วเปลี่ยนค่าก่อนการวิเคราะห์ดังนี้
D=Molecular sequence
O=ANSI
R=1000
Random seed =111
ตอบ Yes จะได้ file ชื่อ outfile ออกมา นำ file ชื่อ infile เก่าเปลี่ยนชื่อแล้ว สร้าง folder ใหม่ 1 folder เพื่อเก็บ file ที่ใช้แล้วไม่ให้มีจำนวน file ใน folder exe มากเกินไป เปลี่ยนชื่อ file outfile เป็น infile เพื่อการวิเคราะห์ในขั้นตอนต่อไปและทำการเปลี่ยนชื่อ file ในรูปแบบนี้ตลอด
8.จากนั้นเปิดโปรแกรม dnadist.exe และตั้งค่าดังนี้
8.1) เปลี่ยนค่า Distance จาก F84 เป็น Kimura 2-parameter โดยการกด D แล้ว Enter
8.2) เปลี่ยนค่า Form matrix จาก Square เป็น Lower-triangular โดนกด L แล้ว Enter
8.3) กำหนด multiple data sets โดยกด M แล้ว Enter จากนั้นโปรแกรมจะถามต้องการใช้ข้อมูลเป็น multiple data sets หรือ weight ให้กด D แล้ว Enter เพื่อเลือก multiple data sets จากนั้นโปรแกรมจะถามจำนวน data sets ในตัวอย่างนี้ซึ่งในขั้นตอนที่ 8 เราเลือกเท่ากับ 1000 จากนั้นกด Enter
8.4) จากนั้น Y แล้ว Enter เพื่อให้โปรแกรมเริ่มทำงาน ซึ่งจะได้ผลลัพธ์เป็น outfile อีกครั้ง

9. เปลี่ยน outfile เป็น infile แล้วคลิก Neighbor เพื่อเข้าสู่การวิเคราะห์การเชื่อมโยงความคล้ายความต่างของลำดับ DNA ที่ได้จากการวิเคราะห์ genetic distance โดยเปลี่ยนค่าก่อนการวิเคราะห์ดังนี้
L =Yes , M = select type D Data set =100, random seed = 111

เมื่อวิเคราะห์เสร็จแล้วจะได้ file 2 file คือ outfile และ outtree แล้ว เปลี่ยน file ชื่อ outfile เป็น infile และ เปลี่ยน outtree เป็น intree
10. คลิกเลือก consense เพื่อการสร้าง Phylogeny Tree เปลี่ยนแปลงค่าดังนี้
T = ANSI

จะได้ file ออกมา 2 file คือ infile และ outtree
11.นำ file outtree คัดลอกไปเปิดใน Treeview program

เมื่อคัดลอกไปแล้วหน้าจอของ Treeview Program เปิดขึ้นแสดงภาพ Phylogeny tree
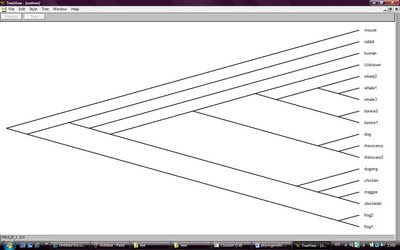

2 ความคิดเห็น:
ตัวเลขที่อยู่ใน phylogenetic tree หรื่อที่อยูบนกิ่งบอกอะไรได้บ้าง
และตัวเลขที่ อยู่ด้านนอกมันบอกอะไรได้บ้างค่ะช่วยตอบหน่อย
ตอบคุณ ยุพาภรณ์
เป็นค่า bootstrapping value ซึ่งมีความหมายว่า จำนวนของกิ่งในแต่ละ tree ที่อยู่ร่วมกันในการทำ bootrapping tree ยกตัวอย่างเช่น ถ้าเราทำ boostripping 1000 ครั่ง แล้วค่าที่อยู่ใน tree เป็นเลข 500 หมายความว่าใน 100 รูปแบบ tree 2 กิ่งนี้ จะอยู่ร่วมกัน 500 ครั่ง
แสดงความคิดเห็น